Datos
Diseñamos la recopilación de datos para que coincida con los puntos de tiempo de decisión en un flujo de trabajo clínico para FET. Por lo general, un embrión se descongelaría a las 0 h (0 h) y se evaluaría su supervivencia inicial y se transferiría a las 2 h (2 h) o 3 h (3 h) después de la descongelación. Sin embargo, su supervivencia sostenida después de este punto de tiempo no puede evaluarse in vivo dado que se ha transferido; solo se puede evaluar el resultado del embarazo. En nuestro diseño, mantuvimos el tiempo de descongelación y evaluación de un flujo de trabajo clínico y determinamos la supervivencia sostenida del embrión en cultivo a las 25 h (25 h) después del proceso de congelación y descongelación. Todos los embriones se cultivaron en medios Global + 10% LGPS (Life Global Protein Supplement), que es el estándar de nuestro centro. Se eligió la supervivencia a las 25 h post descongelación ya que los embriones que no cumplían criterios para congelar o transferir en el día 5 y 6 se cultivan un día más en nuestro centro.
Nuestro resultado primario fue la supervivencia del embrión después de la descongelación a las 25 h. En este estudio, la supervivencia a las 25 h después de la descongelación se definió como la reexpansión de la cavidad del blastocele, la lisis celular mínima y la pulsación con un volumen de blastocele progresivamente mayor. Los embriones descongelados que no se volvieron a expandir, exhibieron volúmenes progresivamente más pequeños mientras pulsaban o tuvieron lisis de más del 50% de las células a las 25 h después de la descongelación se definieron como que no sobrevivieron.
El conjunto de datos consistió en 652 videos de lapso de tiempo de blastocistos posteriores a la descongelación recopilados en el Centro de Salud Reproductiva de la Universidad de California, San Francisco, entre enero de 2019 y mayo de 2020 de 119 pacientes que se ofrecieron como voluntarios para donar sus embriones. Los embriones del estudio fueron previamente biopsiados y colapsados antes de la criopreservación, y consistieron en embriones cromosómicamente anormales donados para investigación. Estas decisiones de disposición fueron tomadas por escrito por los pacientes antes de la crioconservación y las pruebas genéticas. Las imágenes se capturaron utilizando el sistema de lapso de tiempo Embryoscope (Vitrolife, Suecia). Cada video se grabó a partir de la descongelación del embrión, con una velocidad de fotogramas de entre 0,1 y 0,2 h. Los videos fueron revisados, luego anotados por un embriólogo con el resultado de supervivencia binaria: la etiqueta 1 representaba la supervivencia y la etiqueta 0 representaba la falla en la supervivencia, y el embriólogo determinó la supervivencia del embrión al revisar el video de lapso de tiempo completo. El embriólogo desconocía la predicción del algoritmo o de los otros embriólogos participantes y evaluaba el desarrollo continuo del embrión. No hubo observaciones censuradas en el conjunto de datos.
El conjunto de datos se dividió en conjuntos de entrenamiento, validación y prueba. El conjunto de entrenamiento se usó para optimizar modelos, el conjunto de validación se usó para comparar y elegir modelos, y el conjunto de prueba se usó para evaluar los modelos elegidos. Se evaluó a los embriólogos utilizando el conjunto de prueba para que tanto el algoritmo como los embriólogos se evaluaran con los mismos datos. Primero se muestrearon los conjuntos de prueba y los conjuntos de validación de modo que contuvieran un número aproximadamente igual de embriones que sobrevivieron y los que no. Los videos restantes se incluyeron en el conjunto de entrenamiento. No hubo superposición de pacientes entre los videos divididos en cada conjunto.
En este trabajo, nos enfocamos en construir la prueba de concepto demostrando el desarrollo de un algoritmo de aprendizaje profundo y enfocándonos en combinar las predicciones del algoritmo con los expertos. Es importante tener en cuenta que los mapas de prominencia, como los habilitados por el mapeo de activación de clases, serían posibles trabajos futuros que podrían permitir la caracterización de la atención del modelo. Este no ha sido el enfoque de este trabajo y, como tal, no podemos evaluar qué partes de la imagen del embrión está utilizando la computadora para hacer recomendaciones. Además, observamos que las limitaciones recientemente mostradas de los mapas de prominencia ponen en duda su confiabilidad como ayuda para la toma de decisiones.11,12.
Todos los métodos se llevaron a cabo de acuerdo con las directrices y regulaciones pertinentes. El estudio fue aprobado por la junta de revisión institucional (IRB) de la UCSF. Se obtuvo el consentimiento informado de todos los sujetos para el uso de sus tejidos en este estudio.
Modelo de desarrollo
La tarea del algoritmo de aprendizaje profundo era predecir la supervivencia del embrión a las 25 h utilizando imágenes del embrión dentro de las primeras 3 h. Se entrenó un modelo de aprendizaje profundo para generar la probabilidad de supervivencia a 25 h utilizando una sola imagen de un embrión tomada a intervalos de 0,5 h después de la descongelación hasta 3 h. En este estudio, se utilizó una red neuronal convolucional, un tipo particular de modelo de aprendizaje profundo que está especialmente diseñado para manejar datos de imágenes. Las redes neuronales convolucionales escanean una imagen para aprender características de la estructura local y agregan las características locales para hacer una predicción sobre la imagen completa. Los parámetros de cada red se inicializaron con parámetros de una red previamente entrenada en ImageNet. La capa final completamente conectada de la red preentrenada se reemplazó con una nueva capa completamente conectada que producía una salida bidimensional, después de lo cual se aplicó la función softmax. Esta es una función de activación que genera las probabilidades de cada resultado potencial dada una entrada de vector de valor real, donde los valores más grandes corresponden a probabilidades más grandes. En este caso, se utiliza para obtener las probabilidades previstas de éxito y fracaso de supervivencia.
Los modelos fueron entrenados, validados y probados en marcos desde el mismo punto de tiempo posterior al descongelamiento. Antes de ingresar las imágenes en la red, las imágenes se redimensionaron a 224 píxeles por 224 píxeles y se normalizaron en función de la media y la desviación estándar (SD) de las imágenes en el conjunto de entrenamiento de ImageNet. Debido a que el resultado es invariable a las transformaciones de volteo y rotación de la entrada, se aplicó un volteo horizontal aleatorio y un volteo vertical aleatorio con un 50 % de probabilidad a cada imagen en el conjunto de entrenamiento antes de introducirse en el modelo para el aumento de datos; También se aplicó una rotación aleatoria entre 0 y 360 grados.
Todas las variantes del modelo se optimizaron en la pérdida de entropía cruzada binaria mediante el descenso de gradiente estocástico con impulso para actualizar los parámetros del modelo.13. La tasa de aprendizaje se ajustó para cada modelo y estrategia seleccionando la tasa de aprendizaje entre 5e−4, 1e−3 y 2e−3, lo que condujo a la pérdida de validación más baja en el conjunto de validación. La tasa de aprendizaje óptima en todas las arquitecturas modelo fue 1e−3. Para cada modelo y estrategia de entrenamiento, el parámetro de impulso se estableció en 0,9 y la amortiguación se estableció en 0. El parámetro de impulso se utiliza para agregar gradientes en cada iteración para facilitar la convergencia; en este caso, el 90 % del gradiente anterior se suma al 10 % del gradiente actual. Después de cada época (una pasada del conjunto de datos de entrenamiento), el modelo se evaluó en el conjunto de validación y el modelo se guardó en función de la mejor pérdida de validación. Para combatir el sobreajuste, se agregó una caída de peso L2 de 1e-4 a la pérdida de todos los parámetros entrenables. Esto funciona para decaer los parámetros más grandes hacia (pero no exactamente) cero y reduce la posibilidad de sobreajuste del modelo.
Los modelos se entrenaron utilizando cuatro arquitecturas de redes neuronales convolucionales: ResNet18, ResNet34, ResNet50 y DenseNet121, ampliamente utilizadas para la clasificación de imágenes médicas.14,15,16,17. Un conjunto de modelos no propietario, llamado EmbryoNext, se creó promediando las predicciones de clasificación de modelos entrenados con estas diferentes arquitecturas, una metodología común aplicada en el aprendizaje automático. Todos los modelos se entrenaron en GPU NVIDIA GeForce GTX 1070 utilizando la biblioteca PyTorch v1.4.0 utilizando un tamaño de lote de 16 ejemplos.
Puntos de referencia del embriólogo y embriólogo aumentado
Comparamos el rendimiento de EmbryoNeXt con el de embriólogos expertos. Cuatro embriólogos, dos embriólogos junior (2 años de experiencia) y 2 embriólogos senior (más de 8 años de experiencia), calificaron el conjunto de pruebas en una escala de 1 a 10 (con incrementos de 0,5) en tiempos de 2 h y 3 h. La puntuación de 1 a 10 fue una combinación basada en la supervivencia celular (0 a 3), la expansión (0 a 3), la cohesión (0 a 2) y la celularidad (0 a 2) ponderada para favorecer la expansión y la supervivencia celular. Cada embriólogo calificó la probabilidad de supervivencia, examinando primero todos los marcos a las 2 h y luego todos los marcos a las 3 h de forma independiente. Estaban cegados a los verdaderos resultados de supervivencia, historias clínicas e identificadores de pacientes.
También combinamos las predicciones de EmbryoNeXt con las predicciones de cada embriólogo. En esta configuración, el embriólogo fue ‘aumentado’ al promediar las predicciones de rango de EmbryoNeXt con el rango del embriólogo (convertido del puntaje compuesto) en un ejemplo particular, en relación con otros ejemplos en el conjunto de prueba. Se usaron predicciones de rango en lugar de resultados de probabilidad absoluta o puntajes del embriólogo, ya que es posible que tanto los resultados del modelo como los del embriólogo no estén calibrados. En la figura 3 se detalla un esquema de la configuración, el entrenamiento del modelo y la inferencia.
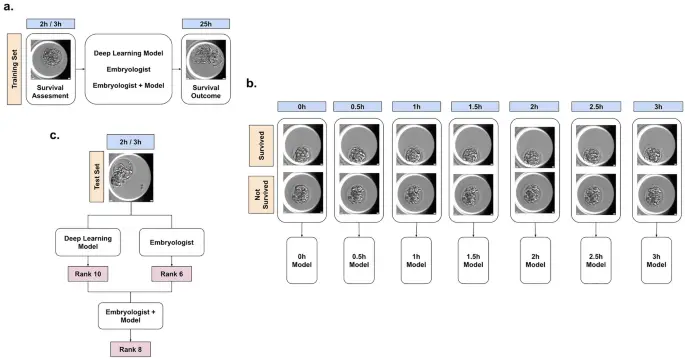
Esquema de la tarea, entrenamiento del modelo e inferencia. (a) La tarea era predecir la supervivencia del blastocisto a las 25 h utilizando una imagen del blastocisto hasta 3 h después de la descongelación. (b) En el momento del entrenamiento, se extrajeron del video fotogramas en diferentes puntos de tiempo de hasta 3 h, y los modelos se entrenaron en ambos videos de embriones sobrevivientes y no sobrevivientes. (C) En el momento de la inferencia, las predicciones del modelo y del embriólogo se convirtieron en rangos de predicción, que se combinaron para producir un resultado de rango de embriólogo aumentado.
análisis estadístico
Comparamos el rendimiento diagnóstico de los modelos y los embriólogos utilizando el área bajo la curva característica operativa del receptor (AUC). Para evaluar si el aumento cambió significativamente el rendimiento, calculamos la diferencia de rendimiento en el conjunto de prueba con y sin aumento. El bootstrap no paramétrico se utilizó para estimar la variabilidad en torno a cada una de las medidas de rendimiento; Se extrajeron 1000 réplicas de arranque del conjunto de prueba, y cada medida de rendimiento y diferencia se calculó para el modelo y el embriólogo en estas mismas 1000 réplicas de arranque. Esto produjo una distribución para cada estimación, y se informaron los intervalos percentiles de arranque del 95 % para evaluar la importancia en el pags= 0,05 nivel. El entrenamiento del modelo y el análisis estadístico se realizaron utilizando Python3.
