Experimentos
El diagrama de flujo de trabajo de este estudio se ilustra en la figura 1. Brevemente, la evaluación se dividió en 3 secciones. Sección 1, la precisión de la autosegmentación basada en DL se evaluó mediante métricas geométricas. Sección 2, la comparación dosimétrica se realizó entre los contornos manuales estándar y los contornos autosegmentados de los planes originales de EBRT. En la sección 3, se exploró el análisis de correlación seguido de métricas geométricas y dosimétricas.
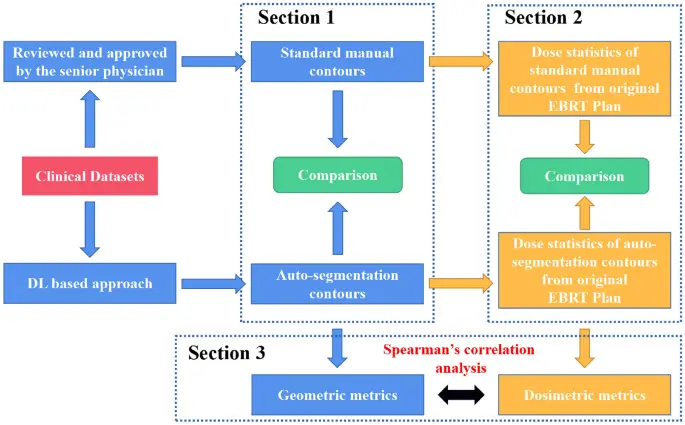
El diagrama de flujo del experimento de evaluación de autosegmentación manual y basado en DL. Los planes EBRT originales se diseñaron y optimizaron en función de los contornos manuales estándar y las estructuras de autosegmentación se transmitieron a los planes EBRT originales para la evaluación dosimétrica.
Conjuntos de datos clínicos
La cohorte independiente de este estudio estuvo compuesta por 75 pacientes con cáncer de cuello uterino que recibieron EBRT en nuestro departamento entre agosto de 2021 y diciembre de 2021. Todas las pacientes fueron diagnosticadas con estadio FIGO IA2-IVB e histología G1-G3, tratadas con una dosis prescrita de 45 Gy- 50,4 Gy (1,8 Gy/fracción). La edad media ± desviación estándar de estos pacientes fue de 55,60 ± 13,35 años. Para cada paciente, se requirió que el agente de contraste se inyectara por vía intravenosa antes de la tomografía computarizada (TC), mientras tanto, las imágenes de la TC se cubrieron desde la parte inferior de la columna lumbar hasta toda la cavidad pélvica y se reconstruyeron con un tamaño de matriz de 512 × 512 y un grosor de corte de 5 mm. utilizando un sistema de tomografía computarizada Philips Brilliance Big Bore (Philips Healthcare, Best, Países Bajos).
La delineación de los CTV de 75 pacientes fue definida manualmente por oncólogos radioterápicos junior, incluidos el cuello uterino completo, el útero, los parametrios bilaterales, la mitad superior de la vagina y los ganglios linfáticos siguiendo las pautas del protocolo del Grupo de oncología radioterápica (RTOG).18. Los OAR relevantes incluidos para los planes de EBRT fueron médula espinal, riñón izquierdo (riñón L), riñón derecho (riñón R), vejiga, cabeza femoral izquierda (cabeza femoral L), cabeza femoral derecha (cabeza femoral R), hueso pélvico, recto y intestino delgado. Las estructuras de planificación de EBRT se realizaron en el Sistema de planificación de tratamiento Pinnacle (Pinnacle, V9.16.2, Philips Corp, Fitchburg, WI, EE. UU.). Todos los contornos manuales fueron revisados y aprobados por oncólogos radioterápicos especializados en cáncer de cuello uterino para generar la delineación estándar.
Segmentación automática basada en aprendizaje profundo
Presentamos un modelo de aprendizaje profundo basado en CNN19 segmentar los CTV y OAR para pacientes con cáncer cervicouterino. Como se muestra en la Fig. 2, la red consta de tres codificadores y tres decodificadores. El InProj se utilizó para extraer las características de la imagen médica, y el OutProj realizó la clasificación por píxeles. Cada codificador y cada decodificador realizaron down-sampling y up-sampling. Todos los filtros de peso de la convolución 2D (Conv2d) tenían un tamaño de ventana de 3 × 3 y un paso de 1. La normalización por lotes (BN) era un proceso mediante el cual sesgaba la distribución de salida y se usaba para la normalización de características. Para esta red, se utilizó la unidad lineal rectificada (ReLu) seguida de cada Conv2d como función de activación de características. Max Pooling podría reducir la cantidad de parámetros y cómputo en la red. ConvTranspose2d era lo opuesto al utilizado para Conv2d, en el que el tamaño de píxel aumenta con un filtro de 3 × 3 píxeles. La conexión de salto se usó para concatenar el codificador y el decodificador del mismo nivel para facilitar la fusión de características multicapa. Utilizamos algunos métodos generales para la mejora de datos (cortar y voltear) para obtener un modelo superior. Este modelo es una arquitectura de segmentación de extremo a extremo que puede predecir etiquetas de clase de píxeles en imágenes CT.
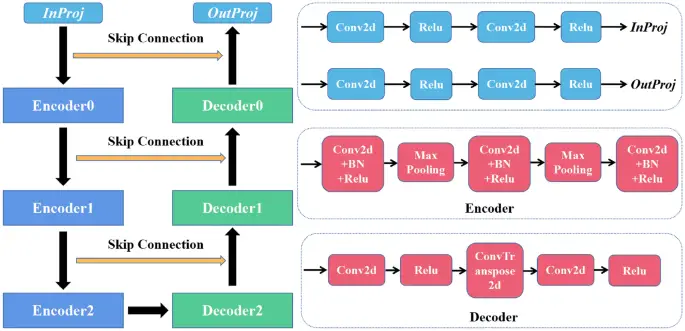
Arquitectura de red de segmentación automática basada en DL.
Se inscribieron un total de 300 tomografías computarizadas clínicas retrospectivas diagnosticadas con cáncer de cuello uterino que recibieron radioterapia para entrenar y validar este modelo, y los conjuntos de datos provinieron de múltiples centros oncológicos para verificar la solidez del modelo CNN. La pérdida de entropía cruzada se seleccionó como la función de pérdida y todos los cálculos de entrenamiento se realizaron utilizando un procesador Intel-Core i7 con una tarjeta gráfica.
Métricas geométricas
La precisión geométrica de los contornos se comparó utilizando el coeficiente de similitud de dados (DSC), la distancia de Hausdorff al 95 % (HD) y el coeficiente de Jaccard (JC). DSC y JC describen la superposición relativa entre la segmentación A y B. HD se utiliza para cuantificar la distancia 3D entre dos superficies de segmentación. El 95% HD es la distancia que indica la mayor separación de superficie a superficie entre el 95% más cercano de los puntos de superficie. Las definiciones son las siguientes:
$$begin{alineado} & DSC = 2izquierda| {A cap B} right|/(left| A right| + left| B right|) \ & HD = max (h(A,B),h(B,A)), ;h(A,B) = mathop {max }limits_{b in B} (mathop {min }limits_{a in A} left| {a – b} right |) \ & JC = izquierda| {A cap B} right|/left| {A taza B} right| \ end{alineado}$$
Para la superposición completa, el valor de HD es 0 y los valores de DSC y JC son 1. Para la superposición incompleta, el valor de HD es grande y los valores de DSC y JC están cerca de 0. Para verificar el rendimiento de reconocimiento del modelo basado en DL en el límite de la segmentación, no se realizó recorte de los bordes superior o inferior para los contornos para este estudio, particularmente en la médula espinal, la cabeza femoral y el hueso pélvico.
Métricas dosimétricas
Los planes de EBRT se calcularon y optimizaron con estos contornos manuales estándar utilizando el sistema de planificación de tratamiento Pinnacle. En la Tabla 1 se presentan las restricciones y métricas dosimétricas. Para CTV, nos enfocamos principalmente en Dsignificar y V100%. Para órganos en serie y órganos paralelos, nos enfocamos principalmente en Dmáximo y Dsignificar, respectivamente. Dsignificar y Dmáximo se definen como la dosis media y la dosis máxima de las estructuras receptoras. V100 se define como el volumen de CTV que recibe el 100% de la dosis prescrita.
análisis estadístico
Se utilizó el software IBM SPSS Statistics (versión 19.0, IBM Inc., Armonk, NY, EE. UU.) y el software Python (versión 3.6.5, Anaconda Inc.) para el análisis estadístico, donde se utilizó la media ± desviación estándar (SD) para presentar y resumiendo los resultados. Para la prueba de concordancia entre los métodos manuales y basados en DL, se utilizó la prueba de Bland-Altman para calcular los límites consistentes para cada estructura de planificación EBRT. PAGS> 0,05 significa concordancia de dos métodos segmentados. Para la diferencia, se realizó la prueba de rangos con signo no paramétrica pareada de Wilcoxon para comparar las variables. PAGS< 0,05 indica que la diferencia es estadísticamente significativa. Las correlaciones entre la métrica geométrica y la diferencia dosimétrica se evaluaron con el análisis de correlación de Spearman.
